生存分析的meta在肿瘤中比较普遍,且比其他的meta分析更容易得到审稿人的青睐。那如何利用其发一篇SCI呢?Revman、Stata、R都可以做生存分析,生存分析比较复杂的是数据的转换,转换过程极其繁琐。此文用meta软件,以HR的转换和合并为例进行讲解。
数据输入与转换

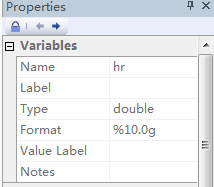
这个是我们需要的数据,我们按照这个数据为例进行处理,输入的时候注意,hr、ll、ul都是数字变量的性质是double,如下图所示:
其余的都是文本数据,变量的性质是str,如图所示:
数据录入完毕后,输入以下代码,完成数据的转换:
gen lnul=ln(ul)
gen lnll=ln(ll)
gen lnhr=ln(hr)
就是对ul、ll、hl取对数
gen selogHR=(ln(ul)-ln(ll))/3.92
计算的是抽样误差均数的标准差
执行的结果是这样的:

数据转换完成后,就可以进行数据的进一步处理了。
数据的处理
这一步可以进行代码操作,也可以进行菜单操作,不过菜单操作太繁琐,使用的时候直接复制我的命令代码到command对话框中就可以。
执行代码:
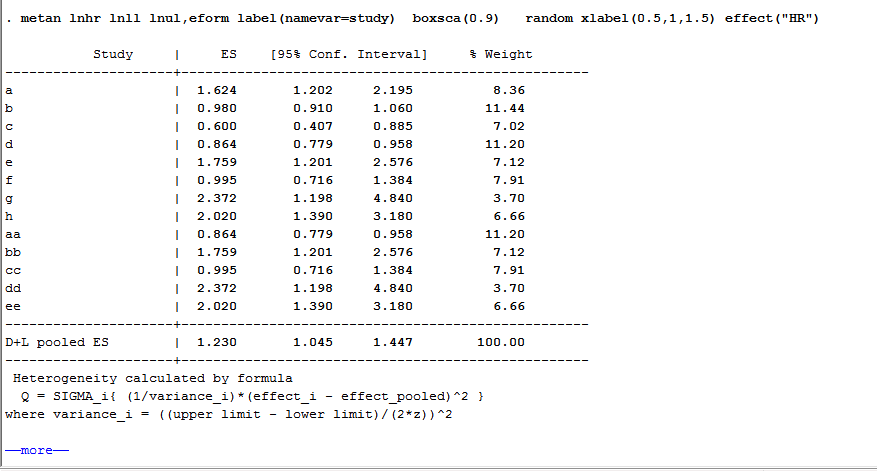
metan lnhr lnll lnul,eform label(namevar=study) boxsca(0.9) random xlabel(0.5,1,1.5) effect("HR")
结果如下图所示:
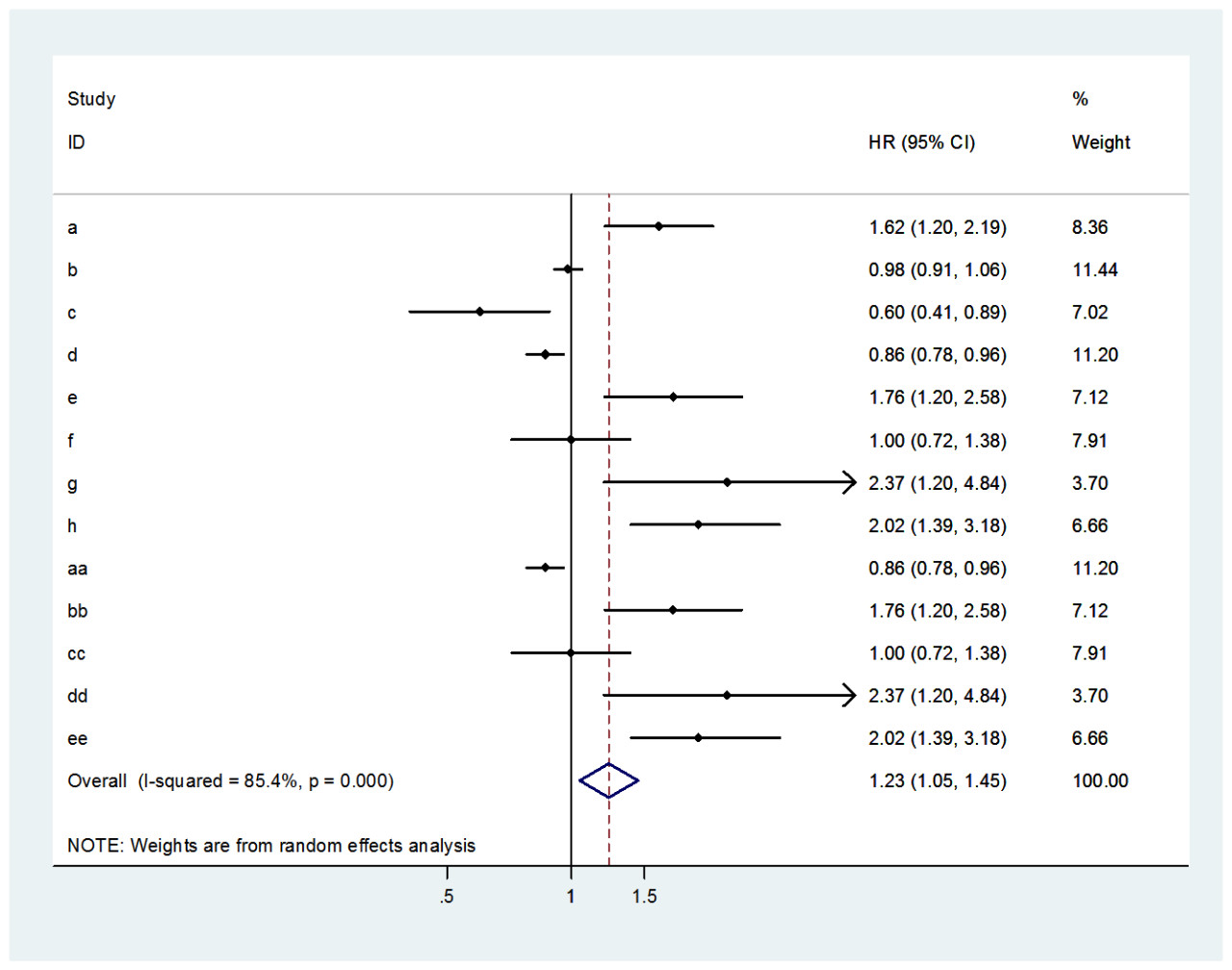
然后再点击一下more就出现森林图了,如下图所示,85%的异质性的确不小,下一步我们进行亚组分析去寻找异质性的来源。
亚组分析
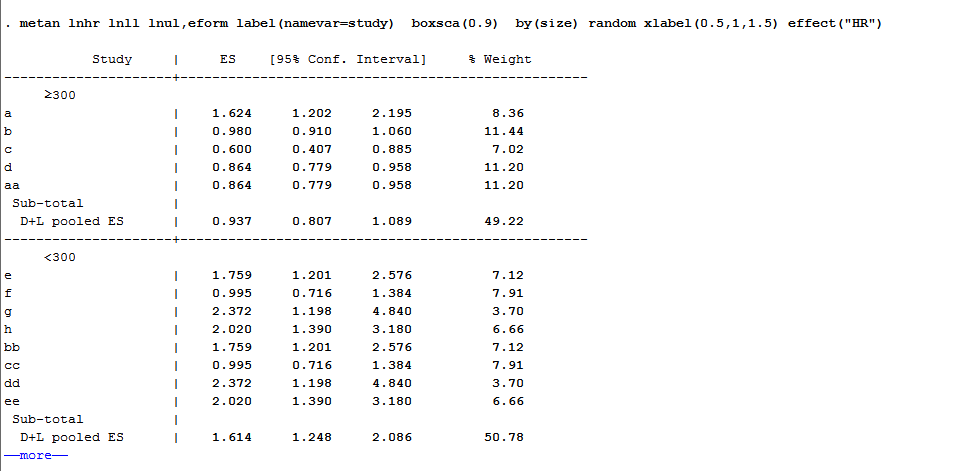
亚组分析我们也用代码去做,因为代码比用菜单操作更加方便,执行代码:metan lnhr lnll lnul,eform label(namevar=study) boxsca(0.9) by(size) random xlabel(0.5,1,1.5) effect("HR")
结果如下图所示:
同样,点击more出现森林图

同样的方法去做cutoff、treatment、country的亚组分析,将 by(size)的size换成cutoff等即可。
发表偏移
执行代码
. metabias lnhr selogHR, graph(begg)
然后结果就出现了
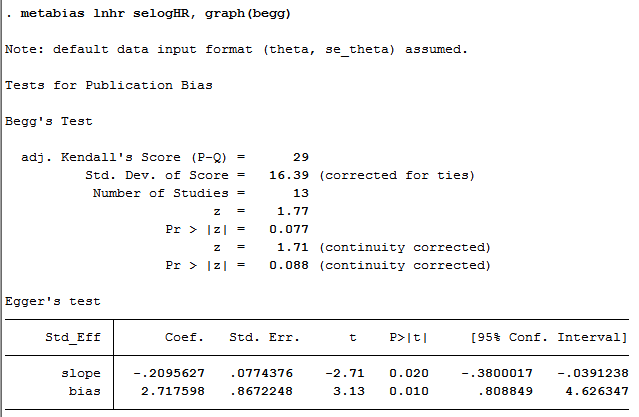
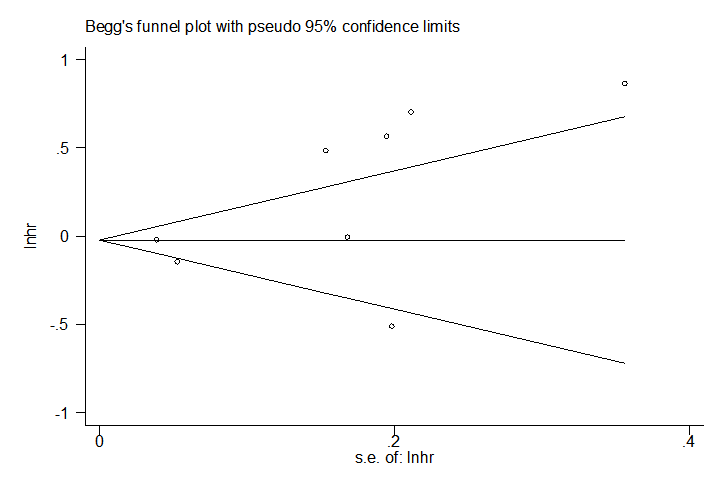
注意了,11个以上的纳入资料才能做发表偏移。再来一个:
执行代码:
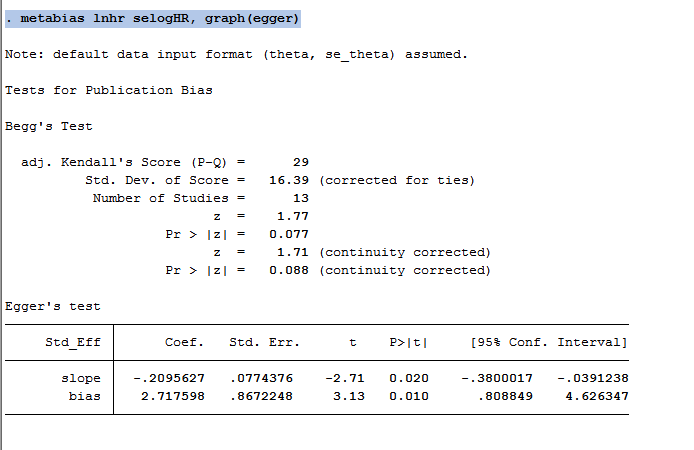
. metabias lnhr selogHR, graph(egger)
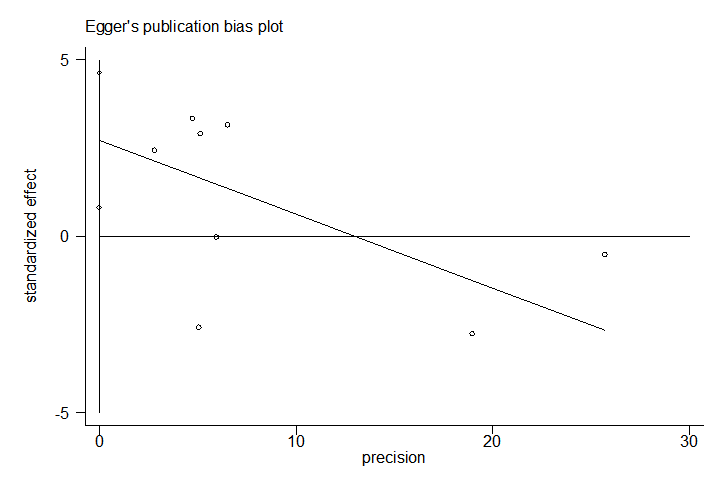
结果如下图所示: