在生信分析中合并表格再常见不过了,因为这些表格是需要进行组合分析的。
下面将根据mRNA和miRNA为例进行合并,其他的合并也类似,把代码中的文件名修改一下就行。
![]()
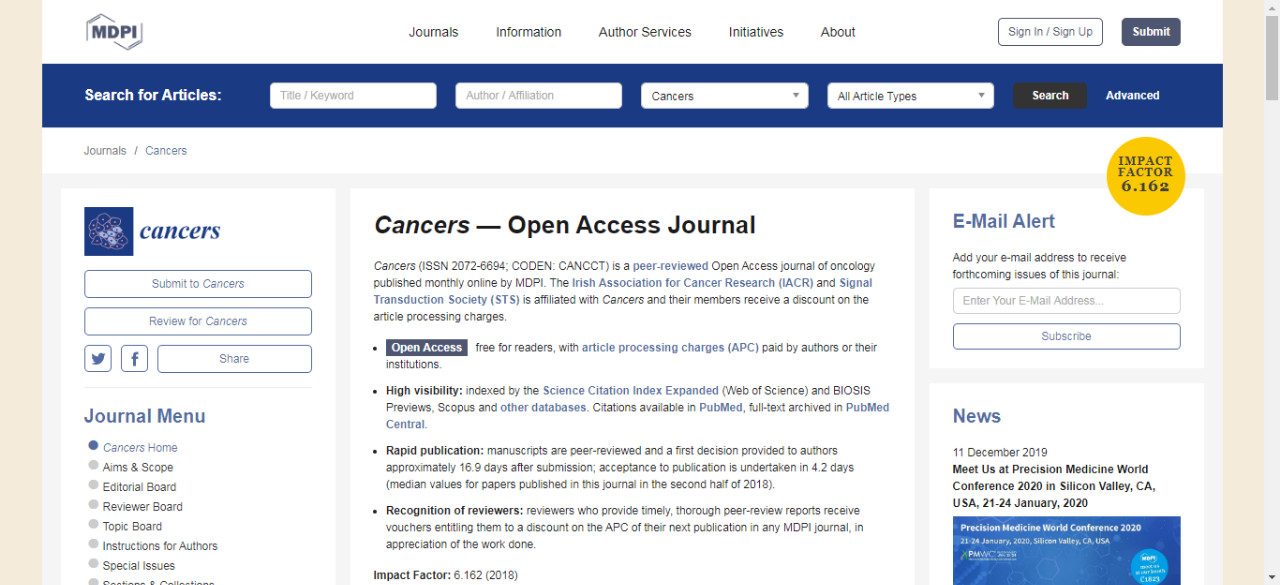
合并前的文件是这样的:
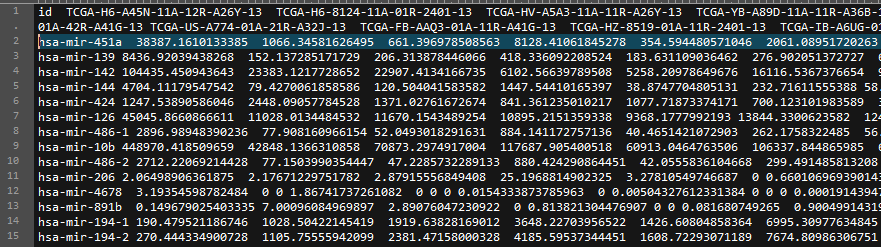
执行代码进行合并:
> setwd("C:\\Users\\dell\\Desktop\\生信学习\\gg") #设置自己的目录,注意了,我们用的是“\\”
> miRNA = read.table("diffmiRNAExp.txt", row.names=1 ,header=T,sep="\t",check.names=F)
> RNA = read.table("diffmRNAExp.txt", row.names=1 ,header=T,sep="\t",check.names=F)
> colnames(miRNA)=gsub("(.*?)\\-(.*?)\\-(.*?)\\-(.*?)\\-.*","\\1\\-\\2\\-\\3\\-\\4",colnames(miRNA))
> colnames(RNA)=gsub("(.*?)\\-(.*?)\\-(.*?)\\-(.*?)\\-.*","\\1\\-\\2\\-\\3\\-\\4",colnames(RNA))
> sameSample=intersect(colnames(miRNA),colnames(RNA))
> merge=rbind(id=sameSample,miRNA[,sameSample],RNA[,sameSample]) #这是合并的过程
> write.table(merge,file="hebing.txt",sep="\t",quote=F,col.names=F) #"hebing.txt"这个生成的合并的文件名,自己修改
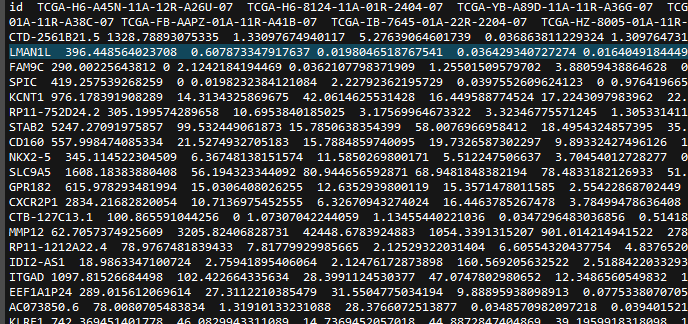
合并后生成文件:
![]()
打开后如下:
两个不同的RNA合并在一起,但是这些混合在一起的RNA来自哪里呢?
下面的这些操作将要帮我们解决这个问题。
执行代码:
> miRNALabel=cbind(rownames(miRNA),"miRNA")
> RNALabel=cbind(rownames(RNA),"gene")
> nodeLabel=rbind(c("ID","Classify"),miRNALabel,RNALabel)
>write.table(nodeLabel,file="qufen.txt",sep="\t",quote=F,col.names=F,row.names=F) #我们把每个基因来源的文件名都给区分,并且生成了文件qufen.txt
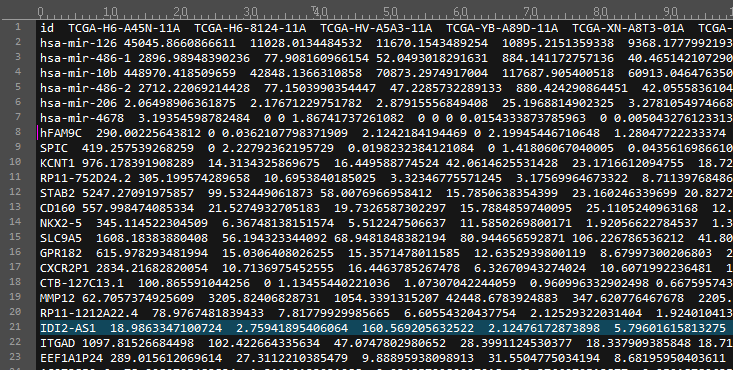
生成的文件是这样的:
![]()
文件打开后是这样的:
我们就可以根据这个文件的信息对RNA的来源进行追根溯源了。
这个过程相对简单一些,将要处理的文件名改为同我的一致,只要复制我的代码,改一下工作目录即可。